Oligo 合成常见问题
Oligo 合成常见问题目录:
- 合成的 Oligo 溶解后发现有少量沉淀,会影响后续实验吗?
- 长度一样的 Oligo DNA 进行 PAGE 电泳时,条带的位置为什么会不一致?
- 合成 Oligo DNA 的 5′ 和 3′ 末端有磷酸基团吗?
- 进行反义核酸实验时,需要对 DNA 链全部进行 S 代修饰吗,除了 S 代外,是否有其他办法能增加核酸的稳定性?
- 双标记荧光探针设计时应遵循哪些原则?
- 荧光探针修饰时,淬灭基团和荧光基团应如何搭配?
- 多重探针检测时应如何选择荧光基团修饰?
- TAMRA、BHQ 淬灭基团应如何选择?
- FITC、6 – FAM、5 – FAM 标记之间有何区别?
- PCR 产物经克隆后测序发现,引物处的碱基有错误,怎么办?
- 平端的 PCR 产物为什么较难克隆?
- 艾科瑞可合成的 DNA / RNA 长度为多少?
OD 值是什么意思?
OD 是 optical density(光密度)的缩写,表示被检测物吸收掉的光密度。
合成的 DNA / RNA 是怎么定量的?
因为核酸在 260 nm 附近有强吸收,因此可根据此性质,用紫外分光光度计对核酸进行定量,用 OD 值表示。1 OD 是指:将 DNA / RNA 溶液置于光路长度为 1 cm 的比色皿中,如果在 260 nm 处其吸光度值为 1,则称 1 ml 该溶液中溶解的 DNA / RNA 的量为 1 OD。1 个 OD 值的合成 DNA / RNA 的质量约为 33 μg(OD 值是总量单位,而不是浓度单位)。
合成 DNA / RNA 的 OD 值如何测定并计算?
将 DNA / RNA 溶液进行适当稀释,使吸光度测定值在仪器允许的精确范围内,再根据原溶液的总体积与稀释倍数计算该溶液的总 OD 值。如,一个 300 μl 的 DNA / RNA 溶液,如果对其进行 5 倍稀释,测定值为 1.0 时,则该溶液的总 OD 值为:0.3 ml × 5 × 1.0 OD / ml = 1.5 OD(若测定值 > 1.5,建议重新稀释后再测定,以保证测定准确性)。
合成的 DNA 干粉如何稀释和保存?
如需长期保存,推荐直接保存干粉或使用无菌 TE 缓冲液(10 mM Tris pH8.0、0.1 M EDTA)溶解成较高浓度的母液(如 100 µM ),于 -20℃ 或 -80℃ 下保存,且应避免反复冻融(不可储存于具有自动除霜功能的冰箱中)。也可用无菌无酶水稀释,或稀释成工作浓度(10 µM),但有效保存时间会缩短。
合成的 RNA 干粉如何稀释和保存?
干粉推荐存放于 -80℃,如不能请于 -20℃ 保存,通常可保存 2 年。溶液状态的 RNA 最好保存在 -80℃,如不行,请保存于 -20℃。
小于 15 mer 的 Oligo DNA / RNA 如何溶解?
Oligo DNA / RNA 长度 ≤15 mer 或序列特殊的情况下,溶解性会降低(Milli-Q 水可能会不溶),建议使用 TE (pH7.5 - 8.0) 溶解。
合成的 Oligos 制品如何进行电泳?
单链 Oligo 容易形成复杂的立体结构,因此不适用于琼脂糖电泳(可能会出现多条带),不宜用 EB、GoldView 或 CelRed 等染色。应使用 15% 变性 PAGE 胶进行电泳,在硅胶荧光板上,紫外灯(254 nm)成像观察。
合成的荧光标记探针如何保存?
荧光探针应严格注意避光保存;干粉于 -20℃ 可保存 2 年不会降解,但应注意避免反复冻融。
合成的 DNA 溶液室温放置几天会降解吗?
一般来说,100 µM 浓度的 Oligo DNA 溶液在常温下(25℃ - 30℃)放置 3~4 天影响不大,但最好不要超过一周。具体时间与合成时器皿、试剂的灭菌程度有关。
Oligo 溶液的 OD260 / OD280 < 1.8,是代表溶液不纯吗?
核酸在 260 nm 附近有强吸收,蛋白质在 280 nm 附近有强吸收,当序列中 A、G、C、T 所占比例大致相同时,可用 OD260 / OD280 比值来评价核酸中是否有蛋白残留 (比值在 1.8~2.2 之间),如从生物体内提取的核酸。但合成的 Oligo 序列很短,其中 A、G、C、T 各种碱基占比不同,由于各种碱基的摩尔消光系数不同,因此不同碱基构成的 Oligo 序列的 OD260 / OD280 比值也不同。例如当序列中 C、T 碱基的含量高时,该比值会大大低于 1.8。此外,序列中碱基的排列顺序也影响该比值。因此无法根据 OD260 / OD280 比值来评价合成 Oligo 的纯度。
| 碱基占比 | OD260 / OD280 |
| 100% A 碱基 | 2.5 |
| 100% G 碱基 | 1.85 |
| 100% C 碱基 | 1.15 |
| 100% T 碱基 | 1.14 |
| A、G、C、T(各 25%) | 1.66 |
Oligo 制品的纯化级别有哪些?区别是什么?
一、脱盐级:简单脱盐,去除合成过程中产生的盐和离子,适合对下游实验要求不太高的较短序列。
二、OPC 纯化级:采用 C18 树脂的小柱进行纯化,可将合成失败的短链 DNA 去除。
三、PAGE 级:根据电荷及分子大小可将目的序列和失败的较长序列进行区分,在一定范围内能有效去除 N-1 杂质链。
四、HPLC 级:通过离子交换柱和反向 C18 柱综合纯化,能更有效地分离 N-1 杂质。
| 纯化方式 | 应用 | 纯化碱基范围 (bases) | 纯度 |
| 脱盐级 | Sequencing、常规 PCR、杂交 DNA 芯片、SNP 分析等 | 10 - 60 | 与序列长度有关 |
| OPC 级 | AFLP、OLA、诊断 PCR、反义核酸、传统修饰等 | 2 - 60 | 80% 以上 |
| PAGE 级 | 分子诊断用引物、定量 PCR、克隆、亚克隆 PCR、基因合成、凝胶迁移评价等 | 10 个以上 | 90% - 95% |
| HPLC 级 | 原位杂交、miRNA、siRNA、定量 PCR、毛细管测序、分子诊断用引物等 | 2 - 60 | 95% 以上 |
| HPLC + PAGE 级 | X-Ray 晶体学研究、NMR、三维结构研究、困难的克隆及基因合成等 | 10 个以上 | 最高纯度 |
合成的 Oligo DNA 制品怎么计算其摩尔数?
由于每个碱基的吸光能力(摩尔消光系数 ε260)不同,较为精确地计算 Oligo DNA nmol 数的公式如下:
nmol 数 = 溶液总 OD 值 * 106 / ε260
ε260=(15.2 * A 碱基数)+(11.5 * G 碱基数)+(7.4 * C 碱基数)+(8.3 * T 碱基数)
合成的 Oligo 溶解后发现有少量沉淀,会影响后续实验吗?
OPC 纯化中,偶而会有微量的 C18 树脂进入制品。树脂不影响任何反应结果,可稍许离心后取上清使用。
长度一样的 Oligo DNA 进行 PAGE 电泳时,条带的位置为什么会不一致?
不同的碱基组成会导致 Oligo DNA 的电泳速度不一致;不同的 DNA 立体结构也会有不同的电泳速度。(通常在较短的序列中发生,长链 Oligo DNA 之间差别较小)。
合成 Oligo DNA 的 5' 和 3' 末端有磷酸基团吗?
若无特殊要求,通常合成的 Oligo DNA 末端不带有磷酸基团,均为 -OH 基。若需要额外加磷酸基团,可在下单时备注说明,此时需收取磷酸化 (PO4 修饰) 的费用。
进行反义核酸实验时,需要对 DNA 链全部进行 S 代修饰吗,除了 S 代外,是否有其他办法能增加核酸的稳定性?
S 代修饰可增加核酸在细胞中的稳定性,避免被核酸酶降解。但不建议整条链全部修饰(注入活的动物体内除外),这样会降低其 Tm 值,也会降低该反义 DNA 与靶序列的结合效率。可采用将 DNA 片段两端插入若干个(通常 3 个)S代磷酯键(Phosphorothioated Bonds),这样既能增加 DNA 的稳定性,又能增加反义 DNA 与靶序列的结合能力。
除 S 代修饰外,2' - OMe - RNA 也可用来增加稳定性,与 S 代 DNA 构成嵌合的反义核酸,既能增强反义核酸的稳定性,又能增加与靶序列的亲和性 (2' - OMe - RNA 与 RNA 的亲和力要比 DNA 与 RNA 的亲和力大很多)。此外,5 - Me - dC 由于能增强 DNA 双螺旋的稳定性,也可被加到 S 代的反义核酸中。
双标记荧光探针设计时应遵循哪些原则?
● 探针应位于两引物之间,可以是正向探针,也可以是反向探针;
● 探针中碱基 G、C 的含量最好能控制在 40%~60% 之间;避免同种碱基成串出现,特别是碱基 G;
● 碱基 G 不要出现在 5' 端;探针的 Tm 值要比引物的 Tm 值高 8~10℃,最好在 68~70℃ 之间;
● 探针长度超过 30 个碱基时,建议把淬灭基团放在中间,以防止荧光本底过高,这时探针的 3' 末端应加磷酸基封阻,以防探针在 PCR 反应过程中延伸。
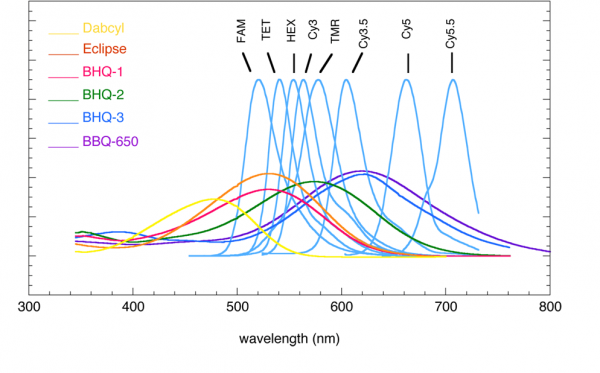
荧光探针修饰时,淬灭基团和荧光基团应如何搭配?
淬灭基团的作用是淬灭报告基团的荧光,因此淬灭基团的荧光光谱应与报告基团的吸收光谱相近。可参考下图:

多重探针检测时应如何选择荧光基团修饰?
传统的荧光定量 PCR 仪通常为四个荧光检测通道,每个通道检测的荧光信号不同,可参考下表进行搭配:
| 激发波长 | 检测波长 | 荧光标记 | |
| 第一通道 | 450 - 490 nm | 515 - 530 nm | FAM、SYBR Green |
| 第二通道 | 500 - 535 nm | 560 - 580 nm | VIC、HEX、Toe、TAMRA、TET、Cy3 |
| 第三通道 | 555 - 585 nm | 610 - 650 nm | ROX、Texas-Red |
| 第四通道 | 620 - 650 nm | 675 - 730 nm | Cy5 |
TAMRA、BHQ 淬灭基团应如何选择?
TAMRA 本身为荧光染料,自身会在更高波长处发射荧光,因此探针荧光本底(Background)相对较高。而 BHQ 系列为非荧光染料,只淬灭报告基团,自身不发射荧光,探针荧光本底低,信噪比更大,检测灵敏度更高。
TAMRA 的吸收光谱覆盖范围窄,可淬灭的报告基团种类较少,而 BHQ 系列染料的吸收光谱覆盖范围较广,可淬灭的报告基团种类更多。所以多重 PCR 检测中常用 BHQ 系列淬灭基团。
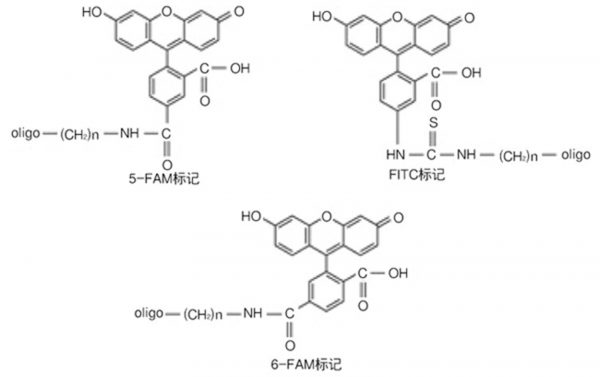
FITC、6-FAM、5-FAM 标记之间有何区别?
三种都是荧光素标记(Fluorescein),发色团均为荧光素;但结构上,5 - FAM 与 6 - FAM 互为异构体;FITC 与 Oligo 的连接方式是硫脲键) ,而前者是酰胺键,通常使用中没有区别。

PCR 产物经克隆后测序发现,引物处的碱基有错误,怎么办?
引物处出现碱基错误,可从以下几个方面进行排查:
● 人为将序列输错(可核对合成报告单);
● 纯化级别是否适合下游实验;
● 重新挑选克隆进行测序(引物纯度不可能是 100%,有可能是挑选了杂质引物扩增出的 PCR 产物的克隆)。
如果通过上述排查仍有问题,艾科瑞将免费重新合成引物。
平端的 PCR 产物为什么较难克隆?
一般的 PCR 引物 5′ 末端没有磷酸基团,因此,扩增后的 PCR 产物的 5′ 末端也没有磷酸基团。当克隆于去磷酸化的末端平滑载体时,无法克隆进去;而当克隆于非去磷酸化的末端平滑载体时,背景会极高。可通过对 PCR 产物的 5′ 端进行磷酸化 (PO4 修饰)处理。
艾科瑞可合成的 DNA / RNA 长度为多少?
合成 DNA 的长度为 1~200 个碱基;合成 RNA 的长度为 1~120 个碱基。所有产品都使用 TOF - MS 进行分子量测定,确保合成的准确性(当合成的 DNA 序列较长时,由于合成及纯化方法的限制,很难保证每个碱基都正确无误)。
